LA STORIA DELLA GENOMICA: COME NASCE, COME SI SVILUPPA E COSA CI RISERVA IN FUTURO
di Sergio Barocci – Università di Genova per la terza età e divulgatore scientifico.
La nuova generazione di tecnologie di sequenziamento del DNA o NGS (parte IV)

Nei primi anni del 2000, diverse piattaforme tecnologiche basate sul sistema di sequenziamento high-throughput incominciarono ad essere sviluppate e continuamente perfezionate.
Queste piattaforme hanno permesso di abbattere notevolmente i tempi di lavoro e i costi di processazione dei campioni, fattori essenziali per favorirne la diffusione. Diverse tecnologie di sequenziamento di 2° generazione sono stati sviluppati da diverse Compagnie e, più tardi, da grandi Gruppi Industriali.
Si differenziano per il tipo di processo biochimico e per i metodi di acquisizione e di elaborazione dei dati ma tutti accomunati da un workflow operativo che prevede tre fasi principali: a) preparazione della sequencing library, b) reazione di amplificazione, c) reazione di sequenziamento.
Next Generation Sequencing
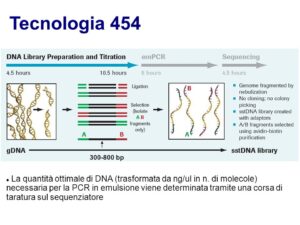
L’era delle cosiddette tecnologie di sequenziamento NGS approda con l’avvento del pirosequenziamento introdotto nel 1996, che si basava sulla misurazione della luminescenza generata a seguito della sintesi di pirofosfati, classificata come sequenziamento ad alto rendimento e implementata in un sistema automatizzato, il sistema 454 della Roche, risultata come la prima piattaforma di sequenziamento di nuova generazione ad arrivare sul mercato.
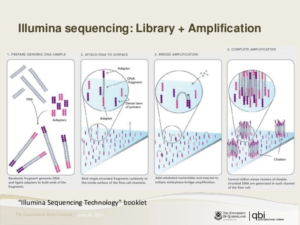
Nel 2007, ci fu una ulteriore svolta nella tecnologia utilizzata per il sequenziamento del il DNA, con lo sviluppo di altre piattaforme degne di nota basate su altre diverse tecnologie come il “sequenziamento per legatura” del sistema SOLiD del 2007 dell’Applied Biosystems e il “sequenziamento mediante sintesi” di Illumina con la piattaforma Genome Analyzer e in seguito la pattaforma MiSeq per piccoli genomi.
sequenziamento di singole molecole

Nel 2009 segue il “sequenziamento di singole molecole” con la piattaforma HeliScope di Helicos un innovativo metodo basato su un sistema ideato nel 2003 dal biofisico israeliano Ido Braslavsky e il sequenziamento SMRT o “single molecule real time sequencing“ con la piattaforma Pac BIO RS II della Pacific Biosciences messa in commercio nel 2013.
Nel 2011 è la volta dello Ion Torrent di Life Technologies che utilizza una tecnologia per rilevare gli ioni idrogeno quando si sintetizza il nuovo DNA, proponendo le piattaforme Ion PGM e Ion Proton.
sistemi portatili di sequenziamento

Nel 2012 vengono invece sviluppati i sistemi di Oxford Nanopore Technologies MinION o Flongle. Si tratta di sistemi portatili per il sequenziamento diretto di DNA e RNA di virus e batteri a cui segue il dispositivo GridION che permette il sequenziamento dell’intero genoma umano, con una piattaforma di supporto in grado di effettuare misure multiple su tante membrane di nanopori (PromethION) per identificare la sequenza nucleotidica.
L’ultima novità di questa Società Tecnologica è rappresentata dalla progettazione di un sequenziatore di dimensioni ulteriormente ridotte “SmidgION” che utilizza la stessa tecnologia di rilvamento dei nanopori di MinION e PromethION, per l’uso con smartphone o altri dispositivi mobili a bassa potenza.
Altre metodologie addizionali
Altre metodologie addizionali, sviluppati in precedenza, comprendono:
a) Polonator sequencing: si tratta di una tecnica di sequenziamento multiplex estremamente accurata utilizzata per leggere milioni di sequenze di DNA immobilizzate in parallelo.
b) DNA nanoball sequening:’ è una originale tecnologia sviluppata dalla Complete Genomics, perfezionata nel 2013 dalla Beijing Genomics Institute (BGI), di sequenziamento ad alto rendimento utilizzata per determinare l’intera sequenza genomica di un organismo. Consiste in un processo di replicazione unidirezionale “rolling circle“ (processo di replicazione unidirezionale dell’acido nucleico in grado di sintetizzare rapidamente copie multiple di molecole circolari di DNA o RNA, come plasmidi, genomi di batteriofagi e genoma circolare di RNA) per amplificare piccoli frammenti di DNA genomico in nanoballs di DNA.
Tecnologie di prima, seconda e terza generazione
 La rapida evoluzione di tutte queste tecnologie NGS , ha reso necessaria una loro classificazione differenziandole in tecnologie di I, di II e di III generazione.
La rapida evoluzione di tutte queste tecnologie NGS , ha reso necessaria una loro classificazione differenziandole in tecnologie di I, di II e di III generazione.
Si tratta di una classificazione non stringente che raggruppa tecnologie di sequenziamento sulla base di caratteristiche peculiari della metodica adottata e sull’ordine cronologico di elaborazione.
Brevemente, rispetto alla metodologia, i sequenziatori di prima generazione si basano sul metodo di Sanger o dei teminatori di catena.
Quelli di seconda generazione si basano principalmente sul pirosequenziamento (Roche/454); sul sequenziamento per sintesi (Illumina/Solexa) con terminatori reversibili fluorescenti; sul sequenziamento per ligazione (Solid/Applied Biosystems) e infine sul rilevamento di ioni idrogeno o H+ (Ion Proton) rilasciati durante il sequenziamento ion semiconductor che, per questa sua peculiarità, è considerata anche di terza generazione insieme ai sistemi a nanopori della Oxford Nanopore e a quelli che sono in grado di sequenziare direttamente singole molecole di acidi nucleici.
piattaforme ngs

Tutte le piattaforme NGS offrono la possibilità di sequenziamento partendo da entrambe le estremità di ogni frammento della libreria e proseguendo in direzioni opposte (paired-end read). In generale, queste reads paired-end forniscono vantaggi che dipendono dalla complessità del genoma e dall’applicazione e dal tipo di esperimento.
Durante questi ultimi anni, le tecnologie NGS si sono notevolmente evolute con l’obiettivo di abbassare i costi, di ridurre i tempi di lavoro e gli steps di processazione, cosi da minimizzare la possibilità di introdurre errori, consentendo la possibilità di eseguire un sequenziamento parallelo e massivo (sino ad un 1 G), a differenza del sistema di Sanger che consentiva il sequenziamento di un solo campione di DNA.
Conclusioni
Oggi, il sequenziamento degli acidi nucleici è diventato parte integrante della genomica moderna. I progressi compiuti nella tecnologia di sequenziamento, dalla sua invenzione agli equivalenti moderni, sono stati davvero straordinari e il 2022 ha segnato il 50° anniversario del sequenziamento del primo gene completo.
L’applicazione delle tecnologie NGS, da alcuni anni, ha potuto aprire nuove frontiere diventando protagoniste in vari settori come la metagenomica, la farmacogenomica e la farmacogenetica, la trascrittomica e l’epigenetica, permettendo di studiare, inoltre, aspetti diversi della cellula, tra cui le mutazioni del DNA, modifiche istoniche, metilazione del DNA, struttura della cromatina e assemblare senza più lacune i genomi completi di tutti i vertebrati. Inoltre, la maggiore conoscenza del DNA umano permetterà di implementare, in futuro, la medicina personalizzata, in primis la terapia genica e l’editing genomico ma soprattutto studi sull’evoluzione e sulla genetica di popolazione.
Ritorno alla prima parte
Bibliografia:
- Binladen J. et al.(2007). “The use of coded PCR primers enables high-throughput sequencing of multiple homolog amplification products by 454 parallel sequencing”. PLoS One. 2007 Feb 14;2(2):e197.
-
Bentley DR. Balasubramanian S,Swerdlow HP. (2008) “Accurate whole human genome sequencing using reversible terminator chemistry.” Nature 2008; 456: 53 – 59
-
Shendure J, Porreca GJ, Reppas NB. (2005) “Accurate Multiplex polony sequencing of an evolved bacterial genome”. Science 309, 1728 – 1732
-
Braslavsky I, Herbert B, Kartalov E. (2003) “Sequence information can be obtained fronm single DNA molecules”. Proc Natl Acad Sci USA. 100, 3960 – 3964
-
Xu M, Fujita D, Hanagata N.(2009) “Perspectives and challenges of emerging single-molecule DNA sequencing technologies.” Small. 23, 2638 – 2649.
-
Eid J. Fehr A. Gray J. et al (2009) “Real Time DNA sequencing from single polymerase molecules “ Science 323(5910): 133.138.
-
Venkatesan BM & Bashir R (2011) . “Nanopore sensors for nucleic acid analysis.” Nat.Nanotechonol. 6, 615 – 624
-
Branton, D. et al. (2008). “The potential and challenges of nanopore sequencing.” Nature biotechnology, 26(10), 1146 – 1153.
-
Jain M., Koren S., Miga K.M. et al. (2018) “Nanopore sequencing and assembly of a human genome with ultra long-reads “ Nat. Biotechnol 36; 338 – 345-
-
Shendure J. et al. ( 2005). “Accurate Multiplex Polony Sequencing of an Evolved Bacterial Genome.” Science 309, 1728 – 1732
-
Porreca, Gregory J (2010). “Genome sequencing on nanoballs“. Nature Biotechnology. 28 , 43 – 44.
-
Nyren P. & Pettersson B. (1993) “Solid phase DNA minisequencing by an enzymatic luminometric inorganic pyrophosphate detection assay.” Anal Biochem 208, 171- 175
-
Ronaghi M., Uhlen M., Nyren P. (1998) “A Sequencing Method Based on Real-Time Pyrophosphate” Science 281, 363 – 365
-
Margulies, M. et al. (2006). “Corrigendum: Genome sequencing in microfabricated high-density picolitre reactors “. Nature, 441, 120 – 121.
-
Myllykangas S., Buenrostro, J., & Ji, H. P. (2012). “Overview of sequencing technology platforms. In Bioinformatics for high throughput sequencing (pp. 11-25).”Springer New York.
-
Shendure, J., & Ji, H. (2008). “Next-generation DNA sequencing”. Nature biotechnology, 26, 1135.
-
Garrido-Cardenas J.A. et al. (2017). “DNA sequencing sensors: An overview.” Sensors 17, 588 – 603
-
Van Dijk, E. L. et al. . (2014). “Ten years of next generation sequencing technology “. Trends in genetics, 30, 418 – 426.
-
Nakano, M. et al. . (2003). “Single-molecule PCR using water-in-oil emulsion” . Journal of biotechnology, 102, 117 – 124.
-
Li, H. et al. . (2016). “CMOS Electrochemical Instrumentation for Biosensor Microsystems: A Review “. Sensors, 17(1), 74.
-
Merriman, B et al. . (2012). “Progress in ion torrent semiconductor chip based sequencing.” Electrophoresis, 33, 3397 – 3417
-
Harris, T. D. et al. (2008). “Single-molecule DNA sequencing of a viral genome”. Science, 320, 106 – 109.
-
Metzker, M. L. (2010). “Sequencing technologies—the next generation”. Nature reviews genetics, 11, 31- 46.
-
Levene, M. J. et al. (2003). “Zero-mode waveguides for single-molecule analysis at high concentrations.” Science, 299, 682 – 686.
-
Branton, D. et al. (2008). ”The potential and challenges of nanopore sequencing.” Nature biotechnology, 26, 1146 – 1153.
-
Eid, J.. et al. . (2009). “Real-time DNA sequencing from single polymerase molecules.” Science, 323, 133 – 138.
-
Solieri, L., Dakal, T. C., & Giudici, P. (2013). “Next-generation sequencing and its potential impact on food microbial genomics”. Annals of microbiology, 63, 21 – 37.
-
Mikheyev, A. S., Tin, M. M. (2014). “A first look at the Oxford Nanopore MinION sequencer.” Molecular ecology resources 14 , 1097 – 1102.
-
Urban, J.. (2015). “Sequencing ultra-long DNA molecules with the Oxford Nanopore MinION.” bioRxiv, 019281.
-
Auton, A. et al. ( 2015). “A global reference for human genetic variation“. Nature. 526 (7571): 68 – 74.
-
Brown C.G. &Clarke J. (2016). “Nanopore development at Oxford Nanopore:” Nat. Biotechnol 34, 810 – 811.
-
Nurk S.Y., Koren S., Rhie A. et al. (2022) ”The complete sequence of a human genome “ Science 376(6588):44-53
