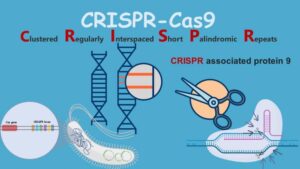
LA STORIA DI CRISPR DALLE ORIGINI
Alla fine degli anni ‘80 e alla metà degli anni ‘90 del secolo scorso, i genomi di diversi lignaggi di batteri e archaea (microrganismi procariotici unicellulari sprovvisti di nucleo e di organuli tipici della cellula eucariotica ma con presenza di vescicole che si originano dalla membrana) rivelarono cluster di brevi ripetizioni palindromiche regolarmente intervallate, conosciute oggi con l’abbreviazione CRISPR. Successivamente si scoprì che queste sequenze ripetute (precedentemente considerate disparate) condividevano un insieme comune di caratteristiche.
BREVI RIPETIZIONI PALINDROME DEL GENOMA
Queste brevi ripetizioni palindrome raggruppate e separate a intervalli regolari, furono scoperte per la prima volta nel 1987 all’Università di Osaka in Escherichia coli dallo scienziato giapponese Yoshizumi Ishino e dal suo team che clonarono accidentalmente una porzione insolita di CRISPR mentre analizzavano un gene (il gene iap) responsabile della conversione dell’isoenzima della fosfatasi alcalina in questo batterio senza conoscerne la funzione.
dal giappone all’europa

Nel 1993, altri ricercatori guidati da Jan D. van Embden nei Paesi Bassi scoprirono che diversi ceppi di Mycobacterium tuberculosis possedevano sequenze distanziatrici diverse tra le ripetizioni del DNA e caratterizzarono questi ceppi in base alle loro sequenze distanziali con un una tecnica nota come “spoligotyping“. Successivamente, queste sequenze furono identificate identificate in molti altri genomi batterici e archeali.
Queste straordinarie scoperte si diffusero in tutto il mondo e tra il 2000 e il 2002 incominciarono ad essere descritte le principali caratteristiche di queste brevi ripetizioni regolarmente distanziate: esse sono costituite da ammassi di brevi sequenze palindromiche ripetute di 20-40 bp separate da sequenze uniche intermedie da 20-60 bp denominate Short Regularly Spaced Repeats o SRSR e successivamente CRISPR. Questi simili cluster di sequenze ripetute e intervallate di informazione genetica provengono da virus e plasmidi che in passato hanno tentato di infettare la cellula.
Proprio queste osservazioni suggerirono un ruolo di CRISPR nell’immunità adattativa dei procarioti.
Short Regularly Spaced Repeats

La ricerca, pubblicata da Francisco J. Mojica nel 2000 e dal suo gruppo all’Università di Alicante ipotizzò un ruolo di CRISPR – Cas nell’immunità adattativa con la presenza di un sistema analogo a quello dell’ RNA – interference (RNAi) presente negli eucarioti cioè che il locus CRISPR era in grado riconoscere gli attacchi da virus esogeni mediante la seguente strategia:
• acquisizione di una sequenza spacer, dovuta a una invasione precedente del virus, nel locus CRISPR del genoma batterico;
• trascrizione del DNA-spacer in RNA;
• utilizzo dell’RNA-spacer per riconoscere la nuova invasione da parte dello stesso virus.
Tale nuova ipotesi si rivelò essere corretta nella maggior parte degli Archei e in una grande percentuale di batteri diversi dall’Escheria Coli.
introduzione del termine crispr
Una migliore comprensione della funzione e dell’importanza di queste sequenze ripetute e intervallate regolarmente si ebbe nel nel 2002 quando Ruud Jansen dell’Università di Utrech, Olanda introdusse il termine CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats o brevi ripetizioni palindromiche raggruppate e separate ad intervalli regolari) per designare queste sequenze raggruppate regolarmente mostrando che sono spesso trovate vicine a una famiglia di geni già descritti in precedenza come un “sistema di riparazione del DNA” e ora chiamati “Cas” (CRISPR-associated) , in grado di codificare per nucleasi ed elicasi, enzimi che tagliano il DNA.
Nel 2005 tre gruppi di ricerca tra loro indipendenti dimostrarono che alcuni spacer o spaziatori presenti nei CRISPR derivavano da piccole sequenze di DNA di batteriofagi o da DNA.
diverse ipotesi sulla funzione di crispr

In parallelo, altre due ipotesi vennero proposte nel tentativo di spiegare la funzione di CRISPR nei procarioti:
• la prima consisteva nell’estensione dell’ipotesi di Mojica cioè del sistema analogo all’RNAi degli eucarioti ai geni Cas e in un’elencazione dei meccanismi d’azione dei diversi sottotipi dei sistemi CRISPR- Cas.
• la seconda ipotizzava che le sequenze CRISPR possono in qualche modo indirizzare gli enzimi CAS a degradare il DNA virale.
Dalle ipotesi di Mojica, negli anni successivi arrivarono le definitive scoperte sulle funzioni principali svolte dal sistema CRISPR – Cas e di conseguenza le sue diverse applicazioni.
Nel 2012, venne pubblicato su Science l’articolo firmato da Jennifer Doudna, biochimica presso l’University di Berkeley in California e da Emmanuelle Charpentier direttrice al Max Plank Institute of Infection Biology di Berlino, entrambe insignite del Premio Nobel per la Chimica del 2020 per lo sviluppo dell’editing genomici CRISPR.
crispr e genoma umano

Nel 2013, durante le prime settimane vennero pubblicati altri tre ulteriori studi per dimostrare la possibilità di utilizzare CRISPR per modificare il genoma di cellule umane (in coltura, in vitro) cellule umane: uno firmato da J. Doudna, uno a firma di George Church dell’Università di Harvard e uno da Feng Zhang dal Broad Institute del MIT di Boston, MA. Queste sono state le pubblicazioni pioniere su CRISPR.
Da questo momento in poi ricerche e sperimentazioni in tutto il mondo hanno migliorato e implementato la tecnica per poterla utilizzare in molti ambiti diversi, dall’agricoltura alla medicina.
Crispr-cas, un sistema immunitario?

Tuttavia, l’origine delle sequenze distanziali rimase sconosciuta e fu solo tre anni dopo che tre gruppi mostrarono indipendentemente che alcune di queste sequenze distanziali hanno un’origine batteriofaga. Nel 2006, Eugene V Koonin e i suoi collaboratori suggerirono che il sistema CRISPR-Cas fosse un sistema immunitario basato sull’interferenza dell’RNA procariotico.
Nel 2007, Rodolphe Barrangou, Philippe Horvath e i loro collaboratori dimostrarono chiaramente che il sistema CRISPR-Cas fornisce un’immunità acquisita contro i batteriofagi: l’infezione porta alla formazione in modo polarizzato di nuove sequenze distanziali e alla protezione contro ulteriori infezioni, che viene abolita con la cancellazione delle sequenze distanziali o di alcuni dei geni Cas.
Negli anni successivi, i meccanismi molecolari furono parzialmente svelati e le caratteristiche strutturali e funzionali delle proteine codificate dai geni Cas descritti, aprendo la strada alla progettazione del sistema di editing CRISPR-Cas9.
metodiche standard di biologia molecolare

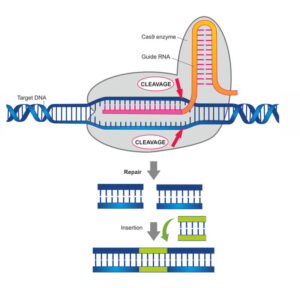
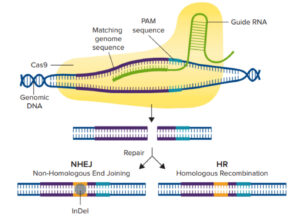
L’attività sperimentale di laboratori in tutto il mondo ha, successivamente, dimostrato che questo sistema è facilmente introducibile in qualsiasi tipo di cellula utilizzando metodiche standard di biologia molecolare. Per effettuare l’editing genomico tramite CRISPR/Cas9 è necessario semplicemente disegnare un RNA guida che riconosca la regione genomica bersaglio. Nel caso in cui si voglia produrre una specifica modifica (es. la riproduzione di una data mutazione) si dovrà anche disegnare e generare un DNA stampo, operazione anch’essa molto semplice e alla portata di laboratori non particolarmente attrezzati e dotati di risorse standard per condurre esperimenti di biologia molecolare.
Oggi, gli scienziati stanno già testando la potenza della tecnologia CRISPR, come viene testimoniato dall’aumento delle pubblicazioni scientifiche.
Alcuni degli esempi più notevoli fino ad oggi sono:
1. Malattie infettive (HIV) e allergie
2. Malattie genetiche (anemia falciforme, fibrosi cistica)
3. Generazione di animali knockout
4. Terapie per i tumori
Lo sviluppo della terapia tumorale utilizzando l’editing genetico è diventato uno degli ultimi obiettivi della tecnologia CRISPR. Modificando le cellule T per rimuovere i geni che possono consentire alle cellule di essere inattivate dalle cellule tumorali, aggiungendo geni che consentono alle cellule immunitarie di colpire meglio i tumori o una combinazione di entrambi gli approcci, si può fornire un modo per sfruttare i meccanismi di difesa del corpo per trattare meglio vari tumori.
Bibliografia:
- Doudna JA and Charpentier E. (2014). “The new frontier of genome engineering with CRISPR-Cas9”. Science 346 (6213),1258096.
- Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A (1987). “Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product“. J Bacteriol. 169 (12): 5429–33
- Jansen R, Embden JD, Gaastra W, Schouls LW.( 2002) .” Identification of genes that are associated with DNA repeats in prokaryotes”. Mol Microbiol 43:1565–1575
- Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier EA.( 2012). “A programmable dual-RNA-guided DNA endonuclease inadaptive bacterial immunity”. Science 337:816–821.
- Le Cong L., Ran F.A., Cox D., Lin S. et al. (2013) .”Multiplex genome engineering using CRISPR/Cas systems” . Science 339: 819 – 823.
- Mali P., Yang L., Esvelt K.M., Aach J. et al. ( 2013). “RNA-guided human genome engineering via Cas 9”. Science 339: 823-826
- Mojica FJ, Díez-Villaseñor C, Soria E, Juez G.( 2000) .”Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria”. Mol Microbiol 36:244–246.
- Mojica FJM, Díez-Villaseñor C, García-Martínez J, Soria E. (2005). “Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements.” J Mol Evol 60:174–182.
RIFERIMENTI:
- https://en.wikipedia.org/wiki/Yoshizumi_Ishino
- https://en.wikipedia.org/wiki/Francisco_Mojica
- https://en.wikipedia.org/wiki/Jennifer_Doudna
- https://en.wikipedia.org/wiki/Emmanuelle_Charpentier
- https://www.nobelprize.org/prizes/chemistry/2020/summary/
- https://en.wikipedia.org/wiki/Feng_Zhang
- https://en.wikipedia.org/wiki/Rodolphe_Barrangou
- https://en.wikipedia.org/wiki/George_Church_(geneticist)
Articolo del prof. Sergio Barocci
Le foto sono estratte da Wikipedia